Forskningsgruppen för medicinska digitala tvillingar
Ett av de viktigaste hälsovårdsproblemen är att många patienter med vanliga sjukdomar som immunologiska, maligna och hjärt-kärlsjukdomar inte svarar på behandling. Detta återspeglar sjukdomarnas komplexitet och heterogenitet. Varje sjukdom kan involvera tusentals gener, som varierar mellan miljarder celler i många organ, såväl som mellan tidpunkter och patienter med samma diagnos. Det finns en stor skillnad mellan denna komplexitet och modern hälsovård.
Vi strävar efter att åtgärda denna skillnad genom digitala tvillingar (DT) av enskilda patienter, vilket nyligen diskuterats av oss i Nature Biotechnology.
DT är datamodeller baserade på klinisk rutin och multiomik-data, ner till enskild cellnivå. DT databehandlas med tusentals läkemedel för att hitta det optimala läkemedlet eller läkemedelskombinationen för enskilda patienter (se figur nedan).
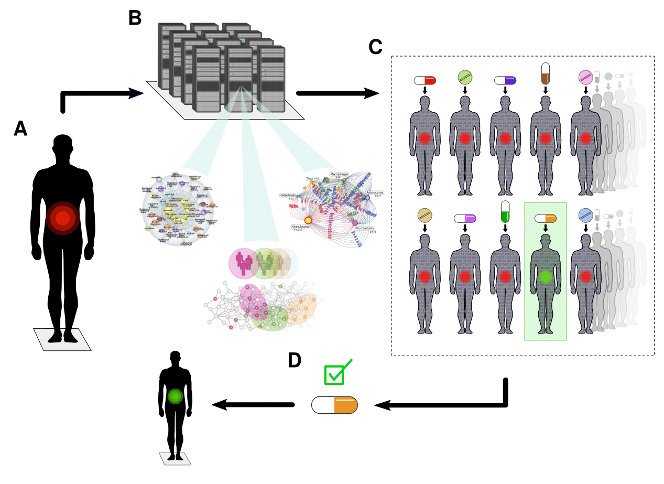
Vårt långsiktiga mål är att varje individ som vill ska erbjudas sin egen DT för tidig förutsägelse och förebyggande av sjukdom. Vår grupp består av kliniska, bioinformatiska, omik- och experimentella forskare, som också ingår i Swedish Digital Twin Consortium.
Besöksaddress
Widerströmska Huset
Tomtebodavägen 18A
171 65 Solna
Postadress
LIME/ Medical Digital Twin Research Group/Plan 4
Tomtebodavägen 18A
171 65 Solna
Vill du veta mer?
Besök gärna sdtc.se, som innehåller länkar till publikationer, inspelade föredrag på Harvard och SciLifeLab, publikationer, TV-intervjuer och en musikvideo skapad av samarbetande artister och en kompositör vid Kungliga Musikhögskolan. Dessutom finns ett inspelat workshop organiserat av US National Academy of sciences engineering and medicine, där SDTC var representerat med ett föredrag.
Gruppmedlemmar
Mikael Benson
Senior ForskareXinxiu Li
ForskningsspecialistForskningsspecialist, ansvarig för klinisk translation.
Hui Wang
Anknuten till ForskningBesökande professor, ansvarig för experimentell validering.
Yelin Zhao
PostdoktorPost doctoral forskare, ansvarig för DT konstruktion och analys.
Jonathan Dorairaj
ForskningsassistentForskningsdataspecialist.
Francesca Borsani
Masters student i precisionsmedicinUtvalda Publikationer
Martin Smelik, Daniel Diaz-Roncero Gonzalez, Xiaojing An, Rakesh Heer, Lars Henningsohn, Xinxiu Li, Hui Wang, Yelin Zhao, Mikael Benson. Combining spatial transcriptomics, pseudotime and machine learning to find biomarkers for prostate cancer. Cancer Research 2025;85(13):2514-2526. doi: 10.1158/0008-5472.CAN-25-0269.
Li X, Loscalzo J, Mahmud AKMF, Aly DM, Rzhetsky A, Zitnik M, Benson M. Digital twins as global learning health and disease models for preventive and personalized medicine.Genome Med. 2025 Feb 7;17(1):11. doi: 10.1186/s13073-025-01435-7
Schäfer S, Smelik M, Sysoev O, Zhao Y, Eklund D, Lilja S, Gustafsson M, Heyn H, Julia A, Kovács IA, Loscalzo J, Marsal S, Zhang H, Li X, Gawel D, Wang H, Benson M. scDrugPrio: A framework for the analysis of single-cell transcriptomics to address multiple problems in precision medicine in immune-mediated inflammatory diseases. Genome Med 2024,
Lilja S, Li X, Lee EJ, Loscalzo J, Zhang H, Zhao Y, Gawel D, Wang H, Benson M. Multi-organ single cell analysis reveals an on/off switch system with potential for personalized treatment of immunological diseases. Cell Reports Medicine 2023:(3):100956. doi: 10.1016/j.xcrm.2023.100956
Li X, Lee EJ, Lilja S, Bohle B, Loscalzo J, Schäfer S, Zhang H, Gustafsson M, Zhao Y, Gawel D, Wang H, Benson M. A dynamic single cell-based framework for digital twins to prioritize disease genes and drug targets. Genome Medicine 2022
Mansour Aly D, Dwivedi OP, Prasad RB, Käräjämäki A, Hjort R, Thangam M, Åkerlund M, Mahajan A, Udler MS, Florez JC, McCarthy MI; Regeneron Genetics Center, Brosnan J, Melander O, Carlsson S, Hansson O, Tuomi T, Groop L, Ahlqvist E. Genome-wide association analyses highlight etiological differences underlying newly defined sub types of diabetes Nat Genet. 2021 Nov;53(11):1534-1542.
Björnsson B, Borrebaeck C, Elander N, Gasslander T, Gawel DR, Gustafsson M, Jörnsten R, Lee EJ, Li X, Lilja S, Martínez-Enguita D, Matussek A, Sandström P, Schäfer S, Stenmarker M, Sun XF, Sysoev O, Zhang H, Benson M; Swedish Digital Twin Consortium. Digital twins to personalize medicine. Genome Medicine 2020;12(1):4.
Jia G, Li Y, Zhang H, Chattopadhyay I, Boeck Jensen A, Blair DR, Davis L, Robinson PN, Dahlén T, Brunak S, Benson M, Edgren G, Cox NJ, Gao X, Rzhetsky A. Estimating heritability and genetic correlations from large health datasets in the absence of genetic data. Nature Communications. 2019;10(1):5508
Lentini M, Lagerwall M, Vikingsson S, Mjoseng HK, Vogt H, Green H, Meehan RR, Benson M*, Nestor CE*. Pervasive experimental errors have severely distorted our understanding of mammalian epigenetics. Nature Methods 2018; 15:499-50.
Shalek AK, Benson M. From bench to bedside: Single-cell RNA-sequencing to personalize medicine. Science Translational Medicine 2017;9(408)
Mika Gustafsson.. Colm E. Nestor, Mikael Benson. A validated gene regulatory network and GWAS identifies early regulators of T-cell associated diseases. Science Translational Medicine 2015;7(313):313ra178
S. Bruhn, Y. Fang, F.Barrenäs, M. Gustafsson,H. Zhang, A Konstantinell, A. Krönke, B. Sönnichsen, A. Bresnick, N. Dulyaninova, H. Wang, Y. Zhao, J. Klingelhöfer, N Ambartsumian, MK, Beck, C. Nestor, E Bona, Z. Xiang, M. Benson. A generally, applicable module-based translational strategy identifies a key diagnostic and therapeutic candidate gene in allergy. Science Translational Medicine 2014:8;6(218)
